NIVELES ESTRUCTURALES DEL ADN
|
|---|
ESTRUCTURA PRIMARIA
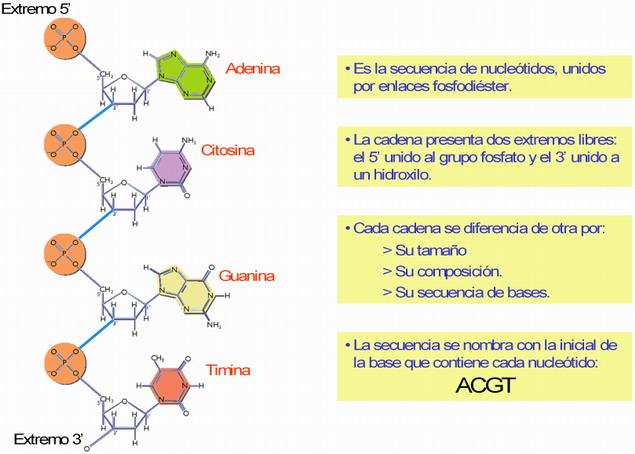
Es la secuencia de nucleótidos unidos por enlaces fosfodiéster (enlaces 5´-3´). Dos cadenas se diferencian por su tamaño, su composición y por la secuencia de bases.
Para indicar la secuencia de una cadena de ADN es suficiente con los nombres de las bases o su inicial (A, T, C, G) en su orden correcto y los extremos 5' y 3' de la cadena nucleotídica.
Ej.: 5'ACGTTTAACGACAAGGACAAGTATTAA3'.
ESTRUCTURA SECUNDARIA
(modelo de Watson y Crick) (modelo de doble hélice)
Datos conocidos hasta WATSON y CRICK (1953):
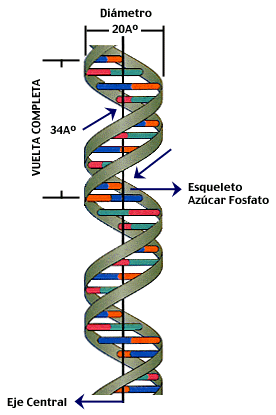
(i) El ADN es una molécula larga y rígida, no plegada.
(ii) Por medio del método analítico de difracción de rayos X, FRANKLIN y WILKINS observaron una estructura fibrilar de 2 nm (20 Å) de diámetro con detalles estructurales repeticidos cada 0,34 nm (3,4 Å) y cada 3,4 nm (34 Å).
(iii) A finales de los años 40 Erwin CHARGAFF vio que la concentración de bases varía de una sp a otra, pero para una misma sp, el contenido de bases púricas (A, T) es igual al de pirimidínicas (G, C). Tejidos dif. de la misma sp tienen la misma composición en bases. Este porcentaje no varía con la edad ni con el estado nutricional ni con las variaciones ambientales.
MODELO DE LA DOBLE HÉLICE (o forma B)
 |
WATSON y CRICK postularon en 1953 un modelo tridimensional para la estructura del ADN que estaba de acuerdo con todos los datos disponibles anteriores: el modelo de doble hélice. Este modelo, además de explicar cómo era el ADN, sugería los mecanismos que explicaban su función biológica y la forma como se replicaba.
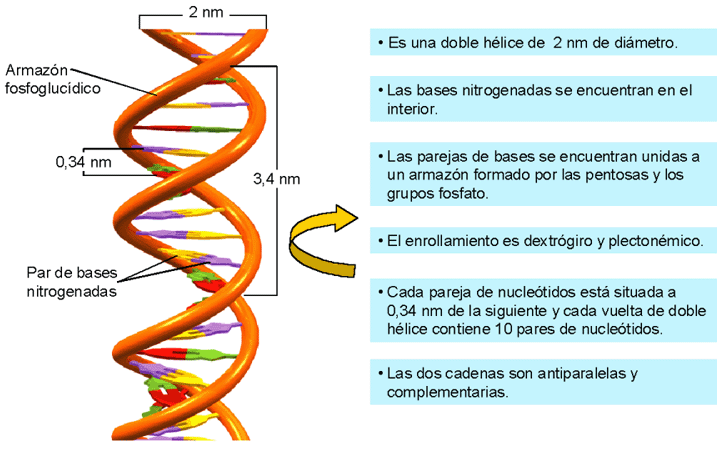
- El ADN estaría constituido por dos cadenas o hebras de polinucleótidos enrolladas helicoidalmente en sentido dextrógiro (dcha.) sobre un mismo eje formando una doble hélice de 2 nm de diámetro según el modelo de la escalera de caracol. El enrollamiento es plectonémico (→ para que las dos cadenas se separen es necesario que se desenrollen).
- Cada pareja de nucleótidos está separada 0,34 nm y cada vuelta son 3,40 nm, ya que está formada por 10 pares de nucleótidos.
- Ambas cadenas son antiparalelas (una iría en sentido 3'→5' y la otra en sentido inverso, 5'→3') y complementarias. |
- Los grupos fosfato estarían hacia el exterior, de forma que sus cargas negativas interaccionarían con los cationes presentes en el nucleoplasma dando más estabilidad a la molécula.
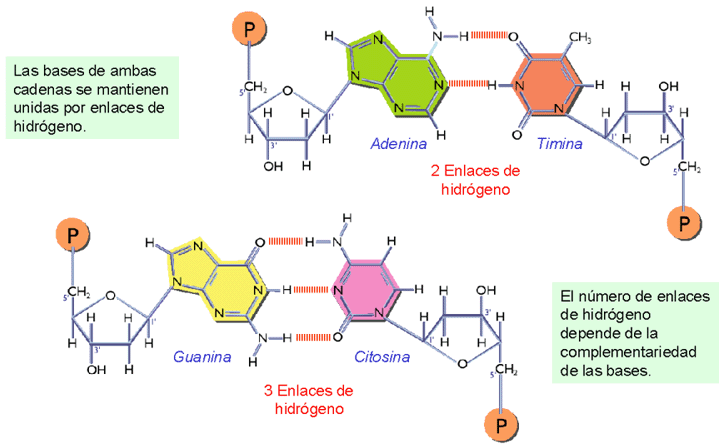
- Las bases nitrogenadas estarían hacia el interior de la hélice con sus planos paralelos entre sí y las bases de cada una de las hélices estarían apareadas con las de la otra asociándose mediante puentes de H entre los grupos NH y los grupos CO.
- El contenido de bases púricas es igual a la de las pirimidínicas, es decir, la proporción de A es igual a la de T, y la de C igual a la de G. Esto indica que el apareamiento se realizaría únicamente entre la A y T, y la G y C, es decir, la complementariedad de las bases A-T y G-C, unidas por 2/3 enlaces de H, respectivamente. (la estructura primaria de una cadena estaría determinada por la de la otra).
| La complementariedad de las cadenas sugiere el mecanismo por el cual el ADN se replica para ser trasferido a las células hijas. Ambas cadenas o hebras se pueden separar parcialmente y servir de molde para la síntesis de una nueva cadena complementaria (síntesis semiconservativa). |
Se han encontrado otras dos configuraciones espaciales del ADN: la forma A y la forma Z.
Forma A del ADN
Se obtiene en lab. por desecación de la forma B. Los pares de bases se encuentran en planos inclinados y el eje de la molécula atraviesa dichos planos por puntos desplazados del centro. Es más ancha y más corta que la forma B.
Forma Z del ADN
Es más larga y estrecha que la forma B. Las dos cadenas de polinucleótidos se encuentran enrolladas formando un zigzag, y es levógira. Se debe a la presencia de numerosos nucleótidos de citosina y guanina alternantes. Puede aparecer en determindas cond. en los seres vivos.
ESTRUCTURA TERCIARIA
(empaquetamiento del ADN) |
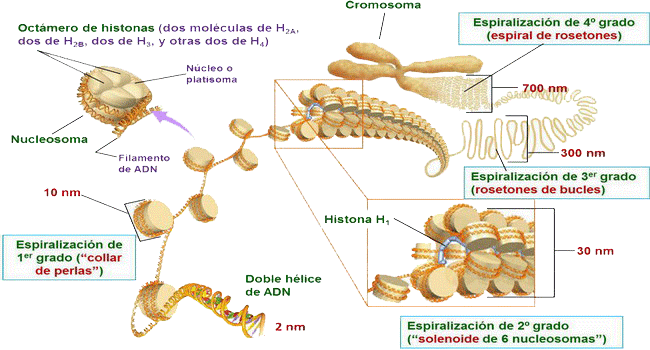
Las grandes moléculas de ADN de las células eucariotas están muy empaquetadas ocupando así menos espacio en el núcleo celular y además como mecanismo para preservar su transcripción.
En las células eucariotas, el ADN se encuentra en el núcleo asociado a ciertas proteínas (→ nucleoproteínas), formando la cromatina. Las proteínas pueden ser histonas y no histonas; las histonas (H1, H2A, H2B, H3, H4) son muy básicas.
| TIPOS DE CROMATINA (en la interfase) |
| Eucromatina |
Es la cromatina menos compacta, ocupando la mayor parte de los espacios intercromatínicos. |
| Heterocromatina |
Es la más compacta y aparece como agrupaciones condensadas teñidas → cromocentros. |
 |
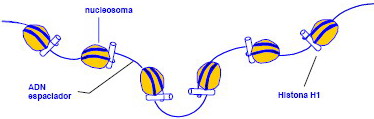
En la cromatina, la doble hélice de ADN se enrolla alrededor de unas moléculas proteicas globulares, las histonas, formando los nucleosomas (10 nm); éstos están unidos por una fibrilla de ADN (2 μm). Cada nucleosoma está formado por un núcleo = platisoma formado por 8 histonas (2H2A, 2H2B, 2H3, 2H4) (excepto H1), y un filamento de ADN (2 μm); la doble hélice de ADN da dos vueltas a su alrededor (200 pares de bases). |
| El conjunto forma una estructura arrosariada llamada collar de perlas |
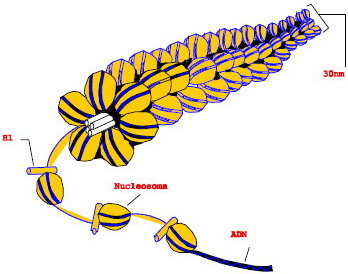
Ahora bien, los nucleosomas pueden empaquetarse formando fibras de un grosor de 30 nm (→ fibra de 30 nm = fibra comatínica o nucleosómica) (espiralización de 1er grado).
Según el modelo del solenoide, las fibras se forman al enrollarse 6 nucleosomas por vuelta alrededor de un eje formado por las histonas H1. |
 |
| SUPERENROLLAMIENTO DEL ADN |
Los siguientes niveles de empaquetamiento no están aún aclarados del todo pero, parece ser que cada fibra se vuelve a enrollar formando un bucle (cada bucle tendría 50 millones de pares de bases), 6 bucles se empaquetarían asociándose a un "esqueleto nuclear" produciéndose un rosetón, 30 rosetones formarían una espiral (300 nm) (espiralización de 2º grado), y 20 espirales formarían una cromátida (700 nm) (superespiralización). Todo ello produciría un gran acortamiento de las largas cadenas de ADN.
| Hacer clic en la imagen para ampliar |
En los espermatozoides el ADN se encuentra aún mucho más empaquetado; se dice que tiene "estructura cristalina".
Los ADN de las bacterias, virus, mitocondrias y plastos no presentan estructuras tan complejas y no están asociados a histonas, aunque sí están asociados a otras proteínas. Ver empequetamiento circular bacteriano.